中国科大实现单根短链DNA分子序列结构化学识别的原理性展示
近日,中国科学技术大学单分子科学团队的董振超研究小组,通过发展与扫描隧道显微镜相结合的亚纳米分辨针尖增强拉曼光谱(TERS)技术,首次在单碱基分辨水平上展示了单根短链DNA分子序列结构的实空间化学识别。该成果于11月27日在国际知名学术期刊《美国化学会志》上在线发表。
生物分子(如DNA、蛋白质等)的序列测定和结构解析对于理解它们的生物学机制和功能至关重要。尽管传统的结构生物学方法已广泛应用于生物分子结构的分析,但对于分子量较小的生物分子,尤其是那些难以标记或结晶的分子,想要在亚分子水平上确定这些生物分子的化学结构仍然存在很大挑战,因此急需开发一种无需标记、且具备高化学灵敏度和高空间分辨率的生物分子结构表征方法。近些年来,单分子针尖增强拉曼光谱技术不断发展,通过把拉曼光谱识别分子结构振动指纹信息的能力和扫描隧道显微镜(STM)的高空间分辨能力结合起来,可以在亚纳米尺度上对单分子的内部化学结构进行实空间表征,为实现生物分子序列结构的识别提供了可能。
2013年,该团队首次在有机小分子体系展示了亚纳米分辨的单分子拉曼成像技术 [Nature 498, 82 (2013)],将具有化学识别能力的空间分辨率提高到了一个纳米以下(~0.5nm)。2019年,团队进一步将空间分辨率提高到了1.5 Å的单个化学键识别水平,并基于这项技术提出了一种重构分子化学结构的新方法 [National Science Review 6,1169(2019)]。然而,对于像DNA链这样的复杂生物分子来说,尽管此前有许多研究组基于大气环境下的TERS技术试图对DNA碱基序列进行识别,但由于来自单个核苷酸的拉曼信号很弱,而且DNA分子的柔性使得测量过程中分子极易发生弯曲和变形,给实验测量带来了巨大挑战,因此至今仍然未能实现单碱基分辨的清晰测序展示,更不用说对单个碱基中的官能团进行识别和定位。
在本工作中,团队以人工设计的单根短链DNA分子作为模型体系,探索TERS技术识别柔性生物分子序列结构的可行性。针对上述挑战,该团队采取了三种策略来攻克难关:(1)通过精确调控STM针尖尖端的原子尺度银团簇结构以构筑等离激元纳腔,使得分子所在的局域电磁场得到高度聚焦和增强,从而实现对DNA分子拉曼信号的增强;(2)通过低温冷却样品(液氮甚至液氦温度)并降低激发拉曼散射的激光功率以提高吸附在表面的DNA分子的稳定性,抑制分子热运动对测量的干扰;(3)通过开发基于电磁脉冲阀的样品制备方法,将溶液中的DNA分子转移到真空下原子级洁净的金属表面,避免DNA分子在衬底表面的聚集和污染,使得清晰的单分子研究成为可能。在此基础上,团队首先对由同种核苷酸组成的短链DNA分子分别进行TERS测量,获得不同核苷酸的拉曼指纹作为参考数据库,然后设计了由四个不同核苷酸组成的单链DNA分子(序列为ATGC)作为原理性展示模型体系,通过STM形貌表征定位单个DNA分子,进一步研究其位置相关的TERS光谱特性,依靠不同核苷酸的光谱指纹特征将各个不同的碱基一一识别出来,从而首次实现在空间和光谱上同时对DNA分子序列结构的清晰识别。通过对单个DNA分子碱基的进一步实空间TERS成像,可以更好地展示这种单碱基水平的分辨能力,从成像图案中不仅可以分辨单个DNA分子中相邻的核苷酸,还可以获得DNA分子吸附构型的重要结构信息,甚至区分碱基和磷酸骨架的空间分布,以及单个碱基T中的甲基基团和单个碱基G/T/C中的羰基基团的空间位置。这些原理性展示结果不仅证明了TERS技术分辨单个碱基、单个官能团的能力,也为跟踪生物分子中的修饰或活性位点提供了可能。展望未来,TERS这种无标记方法如果与阵列纳米加工和人工智能结合起来,有望发展成一种新的系列结构测量技术,为单个生物大分子(如长链DNA/RNA、蛋白质等)序列结构的实空间化学识别提供一种潜在的新途径。
韩彧博士生、董立副研究员和朱路遥博士生为这篇文章的共同第一作者。张超副研究员、张尧教授、董振超教授是文章的共同通讯作者。该系列研究工作得到了基金委、科技部、中国科学院、教育部、安徽省等单位的支持。
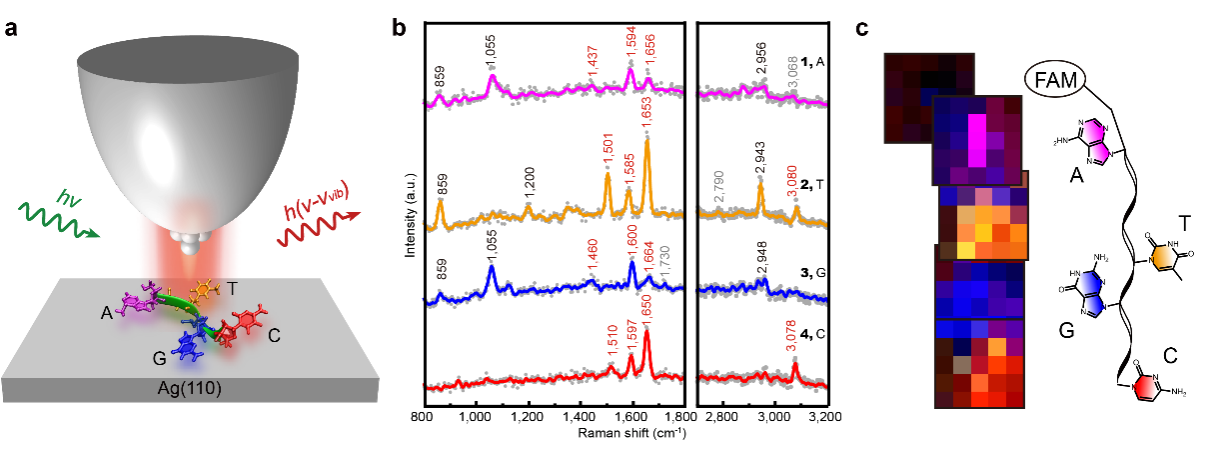
图注:(a) TERS识别单根短链DNA分子序列结构的原理性展示示意图;(b)短链DNA分子中四个碱基上的TERS测量结果,展示出不同碱基的拉曼指纹特征;(c)短链DNA分子的TERS成像以及对应的DNA分子结构示意图。
图注:TERS序列测定的艺术渲染图。(艺术图由董振超教授团队的韩彧、朱路遥、冒文杰设计制作)。
论文链接:https://pubs.acs.org/doi/10.1021/jacs.4c12393
(合肥微尺度物质科学国家研究中心、中国科学院量子信息与量子科技创新研究院、科研部)